搜索


近日,上海交通大学生命科学技术学院微生物代谢全国重点实验室郑舰艇课题组在《Nucleic Acids Research》上发表题为“Structural basis of Streptomyces ECF σShbA factors transcribing principal σHrdB genes”的研究论文。该研究阐明了链霉菌ECF σ因子σShbA识别持家σ因子基因(hrdB)启动子的分子机制,揭示了σShbA通过独特的结构特征调控转录起始。上海交通大学生命科学技术学院博士研究生刘桂扬为第一作者,郑舰艇长聘副教授为通讯作者。
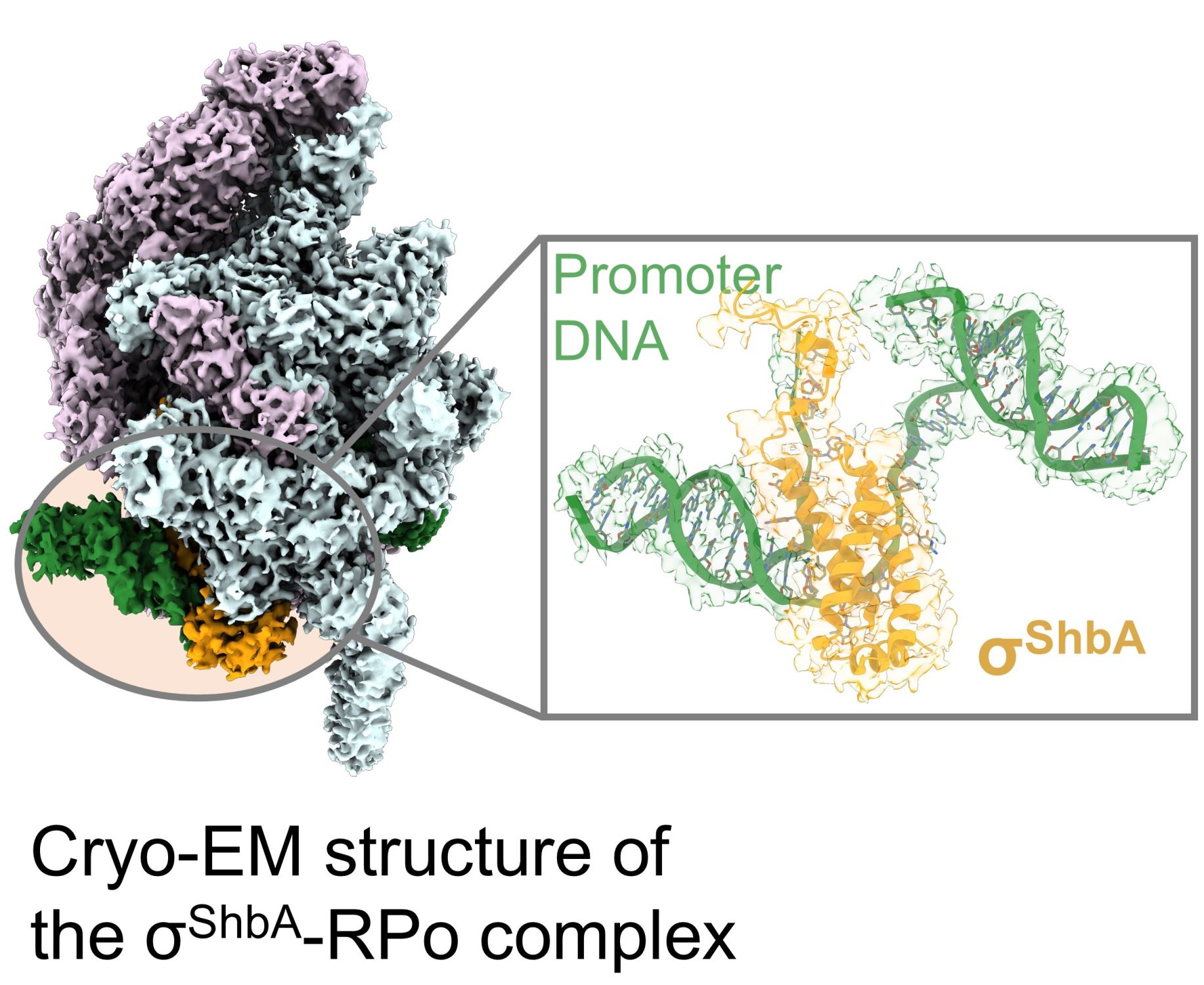
图1. σShbA-RPo的冷冻电镜结构
细菌RNA聚合酶(RNAP)全酶由核心酶和σ因子组成,其中σ因子负责识别启动子序列并起始转录。持家σ因子(如大肠杆菌σ70或链霉菌σHrdB)负责转录维持细胞存活所必需的管家基因,而其自身基因的转录通常由包含持家σ因子的RNAP全酶完成。然而,在模式菌株天蓝色链霉菌(Streptomyces coelicolor)和灰色链霉菌(Streptomyces griseus)中,持家σ因子基因hrdB的转录主要由一种胞外功能(ECF)σ因子σShbA调控,分子机制尚不明确。
研究表明σShbA-RNAP在体外能够转录hrdBp1和shbAp1启动子。冷冻电镜分别解析了σShbA-RNAP全酶与hrdBp1和shbAp1形成的转录开放复合物(RPo)。结构分析表明,σShbA通过其σR2结构域特异性识别启动子的-10区(CGATGA),并形成12 bp的转录泡。σShbA的σR4结构域在复合物中呈现高度柔性,且其缺失对转录活性无显著影响,表明σShbA的转录起始主要依赖σR2。
研究分析了NCBI数据库中所有链霉菌参考基因组,发现σShbA及其调控的hrdB基因在蛋白序列、基因组邻域和启动子保守基序上高度相似。hrdB启动子区域具有CGTAAC-N16-CGATGA基列,而σShbA自身基因的启动子含有CTGCTC-N16-CGCATC基序。两者均包含保守的C-12G-11基序,体外转录表明-12和-11 位碱基的突变降低σShbA的转录效率。
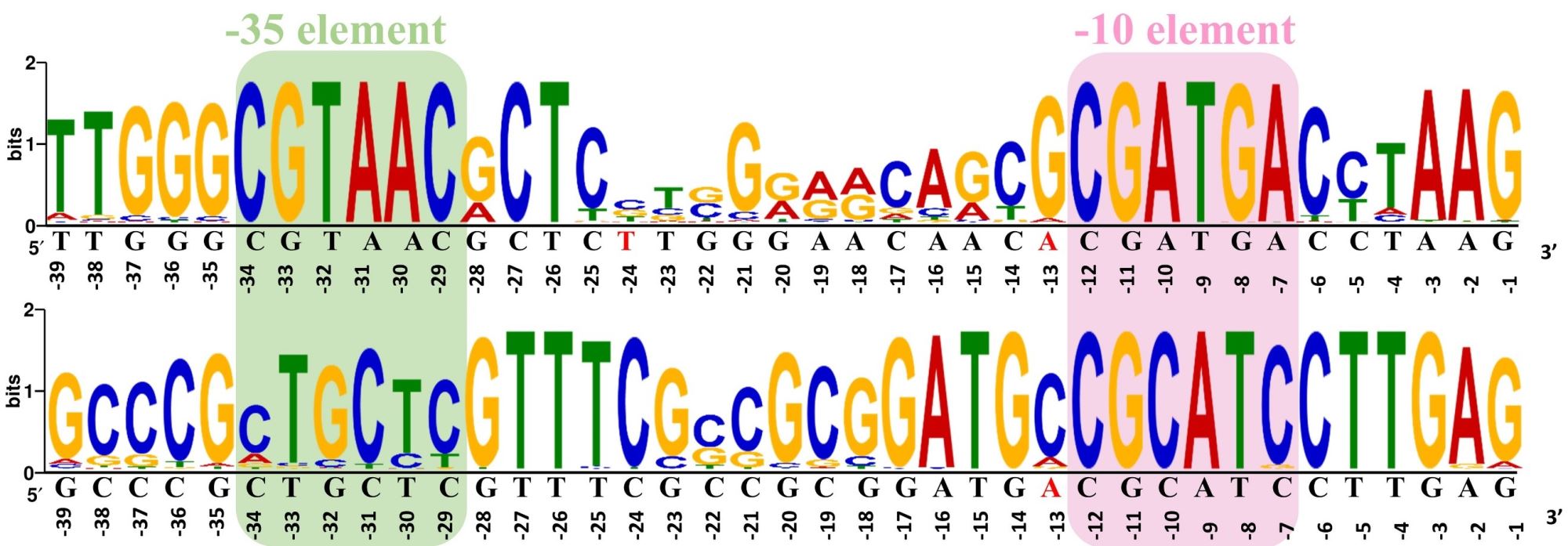
图2. 链霉菌hrdBp1和shbA p1启动子的保守基序
本文结合了结构生物学、生物化学及生物信息学等手段,揭示σShbA调控转录起始的机制与已知的σ因子家族成员(如σ70、σH和σI等)显著不同,拓展了对细菌转录调控多样性的理解,为通过σ因子工程构建链霉菌高产菌株奠定了基础。
本项目得到了国家重点研发计划和国家自然科学基金的资助。感谢上海交通大学分析测试中心和国家蛋白质科学中心(上海)的技术支持。
论文链接:https://doi.org/10.1093/nar/gkaf339


 首页
首页

 学院概况
学院概况

 师资队伍
师资队伍

 人才培养
人才培养

 招生就业
招生就业

 科学研究
科学研究

 平台基地
平台基地

 党群工作
党群工作

 校友之家
校友之家

 安全工作
安全工作

 网上办事
网上办事

 当前位置:
当前位置: